新聞網訊 近日,計算機科學技術學院生物信息研究組研發了微生物組局部比對算法Flex Meta-Storms(FMS),能夠深入挖掘微生物組與其棲息環境之間隱含的聯系。該成果于3月22在線日發表于生物信息學高水平期刊Bioinformatics (SCI IF=6.93)。該研究論文由青島大學獨立完成,計算機科學技術學院為第一單位,碩士研究生張明乾、張文科同學為共同第一作者,學院教授蘇曉泉為通訊作者。該研究獲得了國家重點研發計劃、國家自然科學基金、泰山學者計劃和山東省教育廳的支持,并獲得軟件著作權授權(登記號:2023SR0207831)。
微生物組與周圍的環境有著密切的互作關系。Beta多樣性是微生物組研究與應用的關鍵基礎,能夠定量評估復雜群落之間的差異。當前,beta多樣性主要基于“全局比對”,即利用群落內全部成員計算總體距離(圖1A)。然而,當環境特征(如某些疾病)只與一小部分的微生物有關時,這些微小的改變不足以對整體層面的距離產生影響,從而模糊了微生物與疾病之間的關聯。
針對以上問題,蘇曉泉教授前期提出了微生物組“局部比對”的概念,并應mSystems期刊(SCI IF= 7.324)主編邀請,將此算法概念發表于該期刊的“2021青年科學家特刊”。在此基礎上,研究組首次對微生物組局部比對算法FMS進行了具體實現(圖1B)。與現有的 “全局比對”算法相比,FMS能夠識別出難以感知的細微差異,表現出了更好的敏感性和特異性。因此,FMS算法有助于深入理解微生物組與宿主的相互作用,促進微生物組大數據的深入研究與廣泛應用。
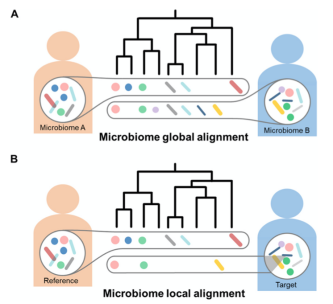
圖1.微生物組beta多樣性距離計算的(A)全局比對模式與(B)局部比對模式
論文信息
1. Mingqian Zhang$, Wenke Zhang$, Yuzhu Chen, Jin Zhao, Shunyao Wu, Xiaoquan Su*, Flex Meta-Storms elucidates the microbiome local beta-diversity under specific phenotypes. Bioinformatics, 2023, DOI: https://doi.org/10.1093/bioinformatics/btad148.
2. Xiaoquan Su*, Elucidating the Beta-Diversity of the Microbiome: from Global Alignment to Local Alignment. mSystems, 2021, DOI: https://doi.org/10.1128/mSystems.00363-21.






![]() 魯公網安備 37021202000856號
魯公網安備 37021202000856號 ![]()